El Premio Nobel de Medicina o Fisiología de 2022 ha recaído en el biólogo Svante Pääbo (Estocolmo, 1955), investigador responsable de distintos descubrimientos sobre el genoma de homínidos extintos, hallazgos que han resultado claves para desentrañar la evolución humana.
Concretamente, el Instituto Karolinska de Estocolmo, institución responsable de la concesión del galardón, le ha reconocido por su contribución decisiva al auge de una nueva disciplina científica: la paleogenómica, que ha permitido revelar las diferencias genéticas entre los humanos actuales y los homínidos desaparecidos y, de esta manera, establecer las bases para explorar qué nos hace humanos únicos.
Como destaca el Instituto, “la Humanidad siempre ha estado intrigada por sus orígenes. ¿De dónde venimos, y cómo estamos relacionados con los que nos precedieron? En definitiva, ¿qué hace que nosotros, el ‘Homo sapiens’, seamos diferentes de otros homínidos?”.
A través de su investigación pionera, Pääbo logró algo que parecía imposible: secuenciar el genoma de un Neanderthal, un ‘pariente’ ya extinto de los actuales humanos. Y también hizo un descubrimiento sensacional de un homínido hasta entonces desconocido: el hombre de Denísova. Pero lo que es más importante, también halló que la transferencia genética que tuvo lugar desde estos homínidos extintos a los ‘Homo sapiens’ ocurrió tras su migración desde África hace cerca de 70.000 años. Y como destaca el Instituto Karolinska, “este antiguo flujo de genes a los humanos actuales tiene relevancia fisiológica, por ejemplo en cómo nuestro sistema inmune reacciona a las infecciones”.
Evolución humana
Los estudios de la evolución humana, básicamente paleontológicos y arqueológicos, evidencian que el ser humano moderno –‘Homo sapiens’– apareció por primera vez en África hace unos 300.000 años, mientras que nuestros ‘parientes’ más cercanos, los neandertales –‘Homo neanderthalensis’–, se desarrolló fuera de África y pobló Europa y el este de Asia hace unos 400.000 años hasta su extinción, ocurrida 30.000 años atrás.
Llegados a este punto, ¿los ‘Homo sapiens’ y los neandertales llegaron a convivir? Pues sí. Hace unos 70.000 años, los ‘Homo sapiens’ migraron de África a Oriente Próximo, desde donde se expandieron al resto del mundo. En consecuencia, coexistieron durante milenios. Pero, ¿qué sabemos de nuestra relación con estos ‘parientes’ ya extintos? Pues las pruebas existentes derivan de la genómica. Y es que a finales de la década de 1990 se logró secuenciar el genoma humano, lo que permitió el estudio de las relaciones genéticas entre las distintas poblaciones humanas. Sin embargo, aún quedaba trabajo por hacer: para ver estas relaciones genéticas con otros homínidos ya extintos, como los neandertales, había que secuenciar el ADN recuperado de especímenes arcaicos.
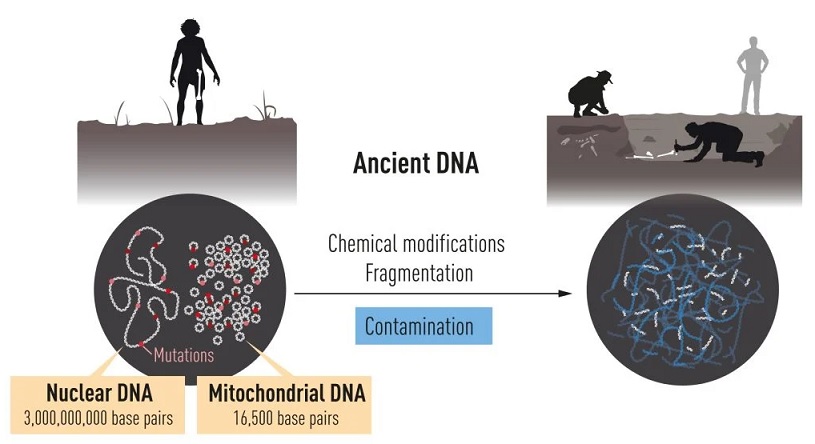
Svante Pääbo, biólogo especializado en genética evolutiva que obtuvo su doctorado en la Universidad de Uppsala (Suecia) en 1986 y que desde 1997 dirige el Departamento de Genética del Instituto Max Planck de Antropología Evolutiva en Leipzig (Alemania), ya buscó en los primeros años de su carrera investigadora utilizar los modernos métodos genéticos para estudiar el ADN de los neandertales. El problema es que con el paso del tiempo el ADN sufre modificaciones químicas y se degrada en pequeños fragmentos. El resultado es que, transcurridos miles de años, solo quedan unos pocos fragmentos de ADN neandertal, que además está masivamente contaminado con ADN bacteriano y de humanos modernos.
Primos no tan lejanos
A principios de los años 90, Pääbo decidió analizar el ADN de las mitocondrias neandertales. El genoma mitocondrial es mucho más pequeño y contiene solo una fracción de la información genética de la célula, pero su disponibilidad es mucho mayor, aumentando las probabilidades de éxito. Así, el investigador logró, por primera vez, secuenciar una región de ADN mitocondrial de un hueso de 40.000 años, y su comparación con los genomas de los humanos modernos y los chimpancés demostró que los neandertales eran genéticamente distintos.
El siguiente paso, una vez mejorada la técnica de aislamiento del ADN de huesos arcaicos, fue analizar el ADN del núcleo celular. Una labor acometida con éxito que le permitió publicar, ya en 2010, el primer genoma secuenciado de un neandertal. Y fruto de este logro, los análisis comparativos demostraron que el ancestro común más reciente de los neandertales y los ‘Homo sapiens’ pobló el mundo hace unos 800.000 años.
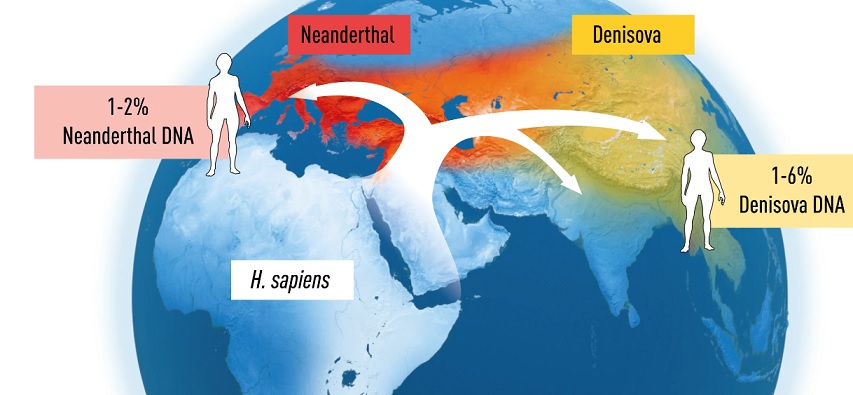
Es más; los estudios también demostraron que el ADN neandertal tiene mayores similitudes con el de los humanos modernos originarios de Europa y Asia que con el de los humanos modernos de África, lo que indica que neandertales y ‘Homo sapiens’ se entrecruzaron durante milenios. De hecho, en torno a un 1-4% del genoma de los humanos modernos de Europa y Asia proviene de los neandertales.
Finalmente, y entre otros hallazgos, el análisis de un hueso del denominado ‘hombre de Denísova’, que vivió hace 40.000 años en el sur de Siberia (Rusia), mostró un genoma diferente del hallado en neandertales y humanos modernos. Pero, una vez más, los análisis comparativos evidenciaron un entrecruzamiento entre el hombre de Denísova y el humano moderno, demostrándose que más de un 6% del genoma de las poblaciones de Melanesia y otras partes del sudeste asiático provienen de este hombre de Denisova.
En definitiva, concluye el Instituto Karolinska, “gracias a estos descubrimientos, ahora entendemos que las secuencias genéticas arcaicas de nuestros parientes extintos influyen en la fisiología de los humanos de hoy. Un ejemplo es la versión ‘denisoviana’ del gen ‘EPAS1’, que confiere una ventaja para la supervivencia en altitud y es común en los tibetanos actuales, o los genes neandertales que afectan a nuestra respuesta inmune a diferentes tipos de infecciones”.